Bogate doświadczenie w badaniach nad PRRSV
Kariera naukowa Dr Hansa Nauwyncka, profesora Uniwersytetu w Gandawie (Belgia) jest naprawdę imponująca. Jest autorem ponad 450 recenzowanych artykułów, 3 rozdziałów w książkach i promotorem 76 doktorató w ciągu ostatnich 30 lat. Uważany jest za jednego z najważniejszych europejskich badaczy, z pewnością najbardziej energicznego! W tym artykule podsumowujemy nasze rozmowy z nim o tym, jak PRRSV ewoluował w tym czasie.

Problemy związane z PRRSV typu 1
Na początku swojej kariery (lata 90.) przeprowadzał eksperymenty w celu zbadania zachowania szczepów PRRSV-1 i PRRSV-2. Chociaż oba wywoływały zaburzenia reprodukcyjne u loch, były one zupełnie inne na poziomie dróg oddechowych u młodych świń. Zaburzenia oddechowe były ewidentne w przypadku typu 2. Szczepy typu 1 powodowały głównie infekcje subkliniczne. W tych latach dowiedział się, że w UE mamy do czynienia z zupełnie innym wirusem niż w USA / Kanadzie. Silna różnica genetyczna była zgodna z wynikami klinicznymi (zdjęcie 1). Doświadczenia terenowe były również bardzo różne. W przypadku szczepów PRRSV-1 wirus rozprzestrzeniał się niezwykle wolno w tuczu. Wirus potrzebował całego okresu tuczu aby zainfekować wszystkie świnie. Wskazuje to na bardzo nieefektywną transmisję. Szybsze rozprzestrzenianie wystąpiło w przypadku PRRSV-2. W Stanach Zjednoczonych naukowcy wykazali rozprzestrzenianie się drogą aerogenną. W pierwszej dekadzie XXI wieku w Rosji / na Białorusi (podtypy 2 i 3 typu 1) zidentyfikowano genetycznie oddalone szczepy PRRSV-1, które były znacznie bardziej zjadliwe i patogenne. Od pewnego czasu podtyp 1 PRRSV-1 rozprzestrzenia się szybciej, a nawet powoduje chorobę kliniczną (szczepy włoskie).
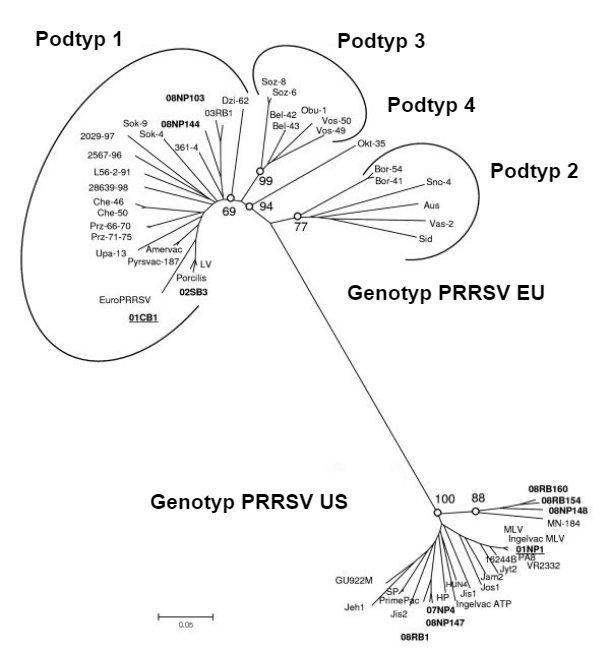
Ryc. 1. Pokrewieństwo filogenetyczne wirusów PRRS, sekwencje ORF 5 ilustrujące różnicę genetyczną między PRRSV typu 1 (genotyp UE) i PRRSV typu 2 (genotyp USA). Źródło Amonsin, A., Kedkovid, R., Puranaveja, S. et al. (2009)
„W Europie Zachodniej nie trzeba się martwić o PRRSV typu 1 jako patogen chorób układu oddechowego; trzeba się bać i zwracać uwagę na towarzyszące mu choroby lub koinfekcje ”- mówi dr Nauwynck. Określił problemy oddechowe związane z PRRS w UE jako „PRRS PLUS” (wprowadził to określenie na weterynarzy terenowych zajmujących się trzodą chlewną) (zdjęcie 1). Liczne koinfekcje PRRSV z różnymi innymi patogenami sprawiają, że PRRSV jest jednym z najważniejszych patogenów w UE. Dlatego niezbędna jest głęboka analiza diagnostyczna. Należy zidentyfikować wszystkie patogeny. W tym kontekście konieczna jest nowa diagnostyka. Pełna wiedza na temat krążących patogenów jest ważna, aby zrozumieć, co dzieje się na fermie i kontrolować PRRS PLUS. Jak mówi - „Jeśli weterynarze zajmą się wszystkimi potencjalnymi koinfekcjami, kliniczne objawy PRRS ze strony układu oddechowego w większości znikną”. Uważa, że europejskie gospodarstwa hodowlane mogą bardzo wydajnie hodować świnie, kontrolując zakażenia współistniejące i nie muszą eliminować wirusa. Może się to oczywiście zmienić, gdy zjadliwość / patogeniczność wirusa wzrośnie w przyszłości.
Ewolucję wirusa można wytłumaczyć lepszą znajomością interakcji wirus-gospodarz; rola receptorów Siglec
His team demonstrated that the virus entry in macrophages involves multiple steps. It starts with the interaction of the virus with siglec receptors and is followed upon internalization with the interaction with the CD163 receptor, leading to disassembly. Different siglecs can be used by PRRSV and evolved in time.
Jego zespół koncentruje się na badaniu interakcji wirus-gospodarz na poziomie molekularnym, dzięki czemu był w stanie zrozumieć, jak wirus zmieniał się w czasie, zwłaszcza jak zwiększał swoją zdolność transmisji, jak opisano powyżej, i jego patogeniczność .
Jego zespół wykazał, że przenikanie wirusa do makrofagów obejmuje wiele etapów. Rozpoczyna się od interakcji wirusa z receptorami siglec, a po internalizacji następuje interakcja z receptorem CD163, prowadząca do rozpadu. Przez PRRSV mogą być wykorzystywane różne receptory siglec, ewoluując w czasie.
Siglec-10 można postawić hipotezę jako „arche-receptor”. Siglec 10 jest obecny na niewielkiej subpopulacji makrofagów w migdałkach i innych tkankach limfoidalnych i jest bardzo skuteczny dla procesu przenikania wirusa, wykazując jego długą koewolucję z PRRSV. Ten siglec był najprawdopodobniej wykorzystywany na samym początku pojawienia się wirusa. W tym czasie PRRSV replikował się tylko w tej małej populacji makrofagów i najprawdopodobniej nie powodował objawów klinicznych. Ponieważ siglec-10 jest również obecny na limfocytach B, PRRSV mógł wiązać się i wnikać, ale nie infekował tych komórek. Może to być podstawą długotrwałości obecności wirusa u jego gospodarza (rezerwuar). Pod koniec lat 80-tych PRRSV najprawdopodobniej przeszedł dodatkowo do receptora Siglec-1 (= sialoadhezyna, CD169) (zdjęcie 2). Komórki Siglec-1 dodatnie są obecne w płucach, łożysku, tkankach limfoidalnych. Replikacja w płucach i łożysku spowodowała problemy z oddychaniem i reprodukcją. Dało to podstawę dla nazwy PRRSV. W XXI wieku wirus w Europie Zachodniej zaczął replikować się w makrofagach nosa, co doprowadziło do wzrostu transmisji między świniami. Nie jest znany receptor odpowiedzialny za tę adaptację; różni się od Siglec-1 i Siglec-10. Wysoce zjadliwy szczep Lena (podtyp 3 PRRSV-1) powoduje uszkodzenia naczyń, co jest związane z replikacją PRRSV w makrofagach żylnych . Receptor w tym podzbiorze makrofagów różni się również od Siglec-1 i Siglec-10. Badane są receptory w makrofagach nosa.
Uważa, że wirus ewoluuje w kierunku form, które infekują coraz więcej subpopulacji makrofagów.
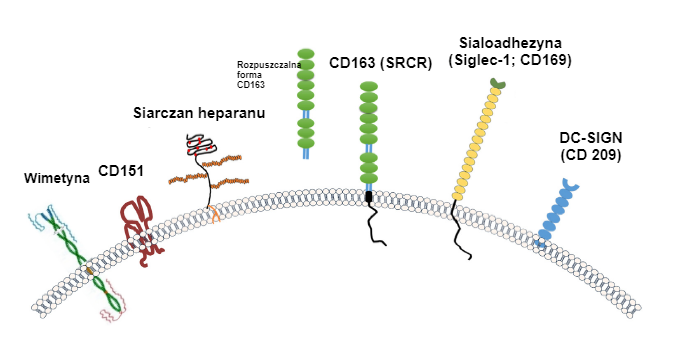
Ryc. 2. Ilustracja receptorów ogólnie uznawanych za związane z PRRSV
Źródło: Zhang, Q., Yoo, D. (2015)








