Rewolucja genomowa jest możliwa dzięki zastosowaniu nowej technologii sekwencjonowania, tzw. "następnej generacji" lub "wysokiej wydajności", które zostały przedstawione w artykule "Co rozumiemy przez rewolucję genomową". Zasadami selekcji genomowej są:

- W celu wykrycia różnic w kodzie genetycznym między zwierzętami, tzw. SNP (Polimorfizm Pojedynczego Nukleotydu), DNA jest analizowane przy pomocy urządzenia "gene chip".
- Poszczególne cechy w danej populacji mają odzwierciedlenie w różnicach w wynikach specyficznych segmentów SNP w DNA.

Jednym z największych długoterminowych potencjałów genomowej selekcji jest określenie konkretnych genów lub regionów genomu ze znacznym wpływem na zdrowie świń, co może być pomocne w określaniu odporności i/lub tolerancji choroby. Kilka dużych programów badawczych pracuje obecnie nad zbadaniem tych obszarów; wciąż rośnie baza danych w literaturze naukowej.
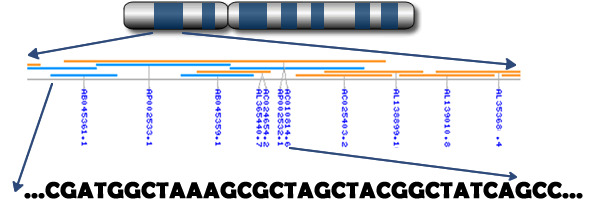
Figure 2. Genomics is identifying SNP’s for health traits (Source: Prof. C. Haley – Roslin Institute, Edinburgh)
Na ostatnim spotkaniu PAG ( International Plant and Animal Genomics), kluczowymi tematami były:
- Naukowcy z USA zidentyfikowali trzy znaczniki, które są istotnie związane ze zmianami w płucach wywołanymi zapaleniem płuc i opłucnej i/lub enzootycznym zapaleniem płuc. Mamy nadzieję, że pozwoli to na identyfikację zmienności genetycznej związanej z poprawą odporności na choroby układu oddechowego.
- Chińscy naukowcy donieśli o różnych mechanizmach chorób między rasą landrace (podatna) i "lokalną" chińską rasą (Tongcheng), która jest odporna na PRRSV. Zaobserwowali inną ekspresję genu CD169 u Tongcheng w porównaniu z Landrace po zakażeniu PRRSV. Niektóre badania wykazały, że receptor CD169 usprawnia prezentację antygenu komórki T.
- PRRS Host Genetics Consortium (PHGC) zostało założone przez grupy badawcze w Kanadzie i USA w celu badania roli genetyki w odporności na zakażenia PRRSV i powiązanych z tym efektów przyrostów. Najnowsze wyniki badań na grupach zwierząt odsadzonych donoszą o zidentyfikowaniu regionu genomu świni na chromosomie 4 (SSC4) mającego znaczący wpływ na zmienność miana wirusa i odpowiedź organizmu. Dalsze analizy i sekwencjonowanie regionu SSC4 będą polegać na identyfikacji markerów, w celu rozróżnienia świń odpornych / z dobrym przyrostem od świń podatnych/ ze słabym przyrostem po zakażeniu PRRSV.
- Dalsze badania PHGC skupiają się na analizie transkryptomu świń zakażonych PRRSV. Dla genów produkujących białka, DNA jest "przepisywanie" na matrycowe RNA (mRNA).Termin transkryptom obejmuje wszystkie mRNA, które są używane do produkcji białek. Badanie to pozwoli rozszyfrować mechanizmy genetyczne uruchamianie w odpowiedzi na infekcję wirusem PRRS.
- Naukowcy z Iowa State donieśli o analizie genomu, które ujawniły wiele różnic w ekspresji genów u świń sklasyfikowanych jako trwali siewcy Salmonelli. Stwierdzili oni, że ilościowe różnice w poziomach IFN-γ wyjaśnia ekspresję większości zbadanych genów. Regulon IFN-γ jest źródłem genów, których poziom ekspresji na dwa dni po zakażeniu ma związek z późniejszym poziomem siewstwa. Takie badania mogą być w przyszłości wykorzystane do opracowania testów predylekcyjnych siewstwa.
- Chińscy badacze badali odpowiedź immunologiczną u prosiąt, badając równocześnie 18 czynników hematologicznych, siedem cech limfocytów oraz trzech cech cytokin po użyciu szczepionki pomoru świń w dwóch genotypach. Zidentyfikowano 12 istotnych SNP, które były skorelowane z odpowiedzią immunologiczną, co daje podstawę dla dalszej identyfikacji mutacji, które leżą u podstaw odpowiedzi ogólnej układu odpornościowego.
- Badacze z Nebraski donieśli o badaniach wykonanych na mieszańcach linii świń, które zostały doświadczalnie zakażone PCV2b wcelu identyfikacji głównych wariantów genetycznych, które wpływają znacząco na wskaźniki postępowania choroby i odpowiedź immunologiczną na PCVAD. W okresie 28 dni zakażenia wykonywano cotygodniowe pomiary przyrostu masy ciała, poziomu wiremii i specyficznych poziomów przeciwciał. Wstępne wyniki dostarczają dowodów na zmienność gospodarza wobec natężenia i czasu odpowiedzi immunologicznej na PCV2b. Większość skupisk SNP, które miały wpływ na wiremię, znajdowały się na kilku chromosomach, w tym: SSC6, SSC7 i SSC12. Region znajdujący się na SSC7 wpływa również na przyrosty masy ciała w czasie zakażenia. Skupiska genów dla SLA klasy II jest również zlokalizowany w tym regionie, co może wyjaśniać obserwowaną zmienność fenotypową.

Figure 3. PCVAD – a key future target for genomic selection (Source: J. Mackinnon)
Oprócz powyższych badań, trwają prace nad genami/ SNP zaangażowanymi w takie jednostki chorobowe jak pryszczyca, afrykański pomór świń, czy świńska grypa. Jednak w przyszłości selekcja dla poprawy zdrowia wymaga zastosowania wielu markerów oraz dostępności dużych zbiorów danych DNA z rodowodowych populacji zwierząt z dokładnie mierzonymi fenotypami, które określą korelację między SNP i cech zdrowotnych. Następny artykuł z tej serii, przyjrzy się temu szczegółowo.