W naszej poprzedniej dyskusji podkreśliliśmy, jak dużym wyzwaniem jest prowadzenie rzetelnych naukowo badań nad mikrobiotą świń. Mimo to obserwujemy coraz większą liczbę badań publikowanych na ten temat (Wykres 1). W miarę jak coraz więcej informacji staje się dostępnych, jak można wyciągnąć sens z tych danych - i zastosować je na fermie?
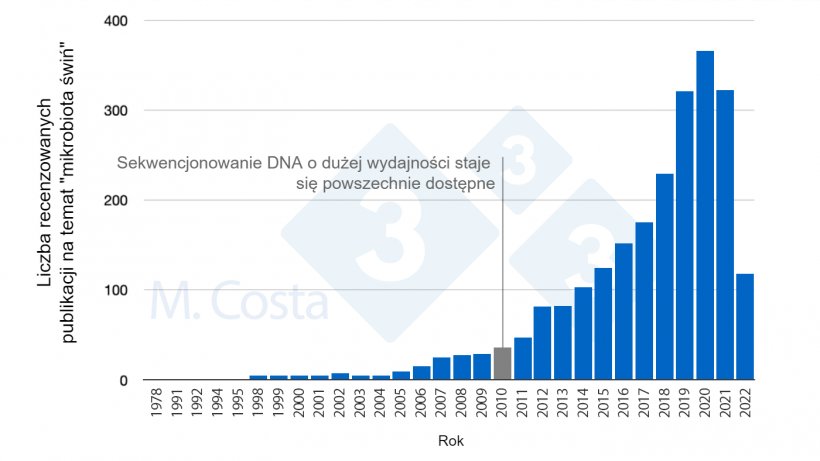
Wykres 1. Liczba recenzowanych badań opublikowanych w ciągu ostatnich 50 lat dotyczących mikrobioty świń.
Krok 1 – Poznaj swoje warunki.
Istnieje kilka kluczowych metryk stosowanych w badaniach mikrobiomu, które ułatwiają nam zrozumienie wyników. Wiele z nich zapożyczono z ekologii "makro" - w końcu społeczność bakterii w jelitach nie różni się wiele od ławicy ryb w morzu. Obie mają relacje ze swoim środowiskiem i innymi istotami, które dzielą z nimi przestrzeń/siedlisko. Dlatego zrozumienie tych metryk pozwoli na właściwą interpretację eksperymentów skoncentrowanych na mikrobiomie. Ogólnie rzecz biorąc, badania takie skupiają się na wyjaśnieniu, jakie mikroby są obecne/nieobecne w danej próbce - odpowiadają na ogólne pytanie "kto tam jest" i "ile was tam jest"? Różne gatunki/rodzaje mikrobów w danym siedlisku (np. jelicie) są często określane jako różnorodność społeczności. Ta różnorodność jednostek (np. owoców, bakterii) w jednym siedlisku (np. kał od świni odsadzonej #495 w 3 dniu po odsadzeniu) jest nazywana różnorodnością alfa (Wykres 2) i bierze pod uwagę dwa główne aspekty:

- Bogactwo gatunkowe: ile jest różnych rodzajów bakterii/gatunków.
- Równomierność gatunkowa: rozmieszczenie (obfitość) tych gatunków, czy jeden organizm dominuje (jest bardziej obfity) nad innymi.
Istnieje wiele indeksów stosowanych do pomiaru różnorodności alfa, z których najbardziej powszechne to indeks Shannona, Chao1 czy Simpsoma. Każdy z nich odzwierciedla inne aspekty różnorodności alfa. Indeksy te można często zobaczyć w badaniach mikrobiomu, odzwierciedlając różnorodność wewnątrz próbki.
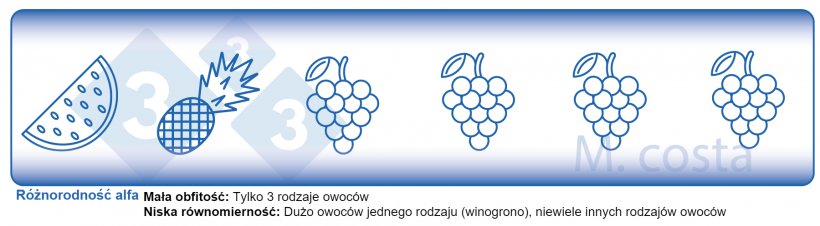
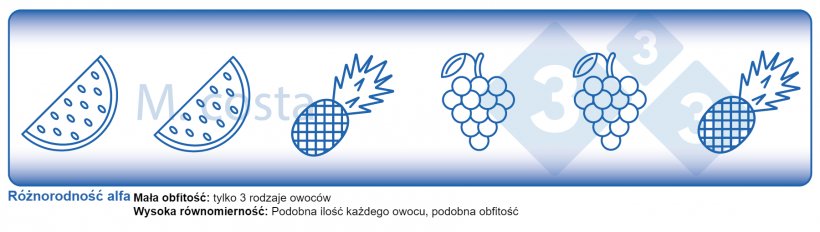
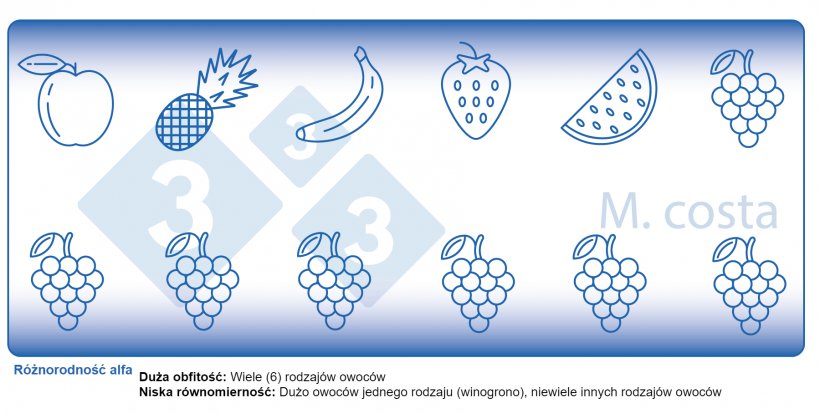

Schemat 2. Jak interpretować miary różnorodności mikrobiologicznej w obrębie próbek (różnorodność alfa). Różnorodność jest produktem bogactwa (np. rodzaje owoców) i równomierności (np. rozmieszczenie lub obfitość każdego rodzaju owoców) w danej próbie.
Porównując społeczności bakteryjne dwóch siedlisk (np. próbki kału świń leczonych i nieleczonych antybiotykami), badamy ich różnorodność beta - podobieństwo między próbkami. Czy te społeczności mają wiele wspólnych cech (np. czy możemy znaleźć te same bakterie w próbce kału A i B, Ryc. 3)? Czy są one podobne czy niepodobne? Ponownie, istnieje wiele indeksów używanych do pomiaru tego, takich jak Unifrac, Jaccard, Bray-curtis dissimilarity, itp. Uwzględniają one różne aspekty różnorodności społeczności, takie jak (fito)genetyczne relacje między mikrobami lub ich liczebność w każdej próbce. Schemat 3. Zrozumienie zmian w składzie mikrobów pomiędzy próbkami (różnorodność beta). Bardziej podobna społeczność mikrobiologiczna dzieli więcej typów mikrobów pomiędzy próbkami. Zazwyczaj przy obliczaniu wskaźnika różnorodności beta bierze się pod uwagę inne aspekty (takie jak związki genetyczne między mikrobami).
Ogólnie rzecz biorąc, różnorodność alfa mikrobów jelitowych ma tendencję do zwiększania się, a różnorodność beta ma tendencję do zmniejszania się w ciągu życia świni (rysunek 4).
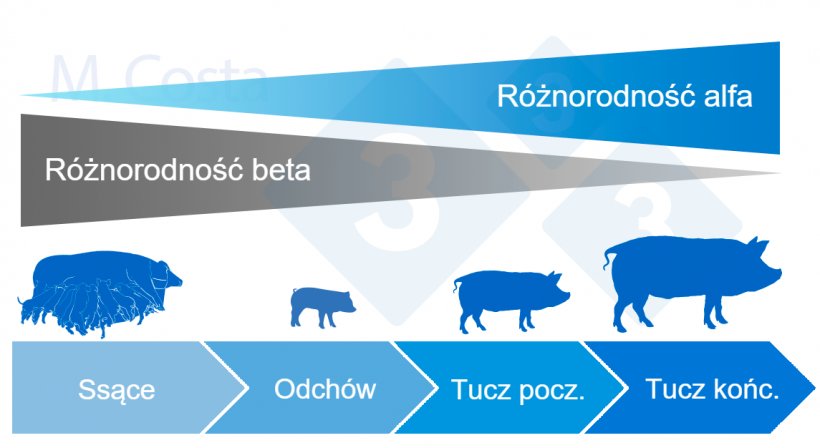
Schemat 4. Dynamika zróżnicowania alfa i beta w ciągu życia świni.
Krok 2 – Czy badanie obejmowało odpowiednie kontrole?
Jeśli chodzi o badania mikrobiomu, należy dołożyć wszelkich starań, aby zminimalizować wpływ zanieczyszczeń. Mikrobowe DNA jest dosłownie wszędzie (nawet jeśli mikroby są martwe, ich DNA tam jest), a obecne technologie sekwencjonowania DNA mogą wykryć bardzo małe jego ilości (jak omówiono w części 1). Dlatego badania nie tylko muszą obejmować odpowiednie grupy doświadczalne (np. świnie leczone lub nie leczone antybiotykami w tej samej partii, jedzące tę samą dietę, itd...), ale także odpowiednie kontrole techniczne (puste probówki do pobierania próbek, zestawy do sekwencjonowania bez próbek), aby pomóc zidentyfikować zanieczyszczenia i usunąć je z analiz. Jeśli nie zostaną one ujawnione w badaniu, wszelkie wnioski mogą być obarczone błędem.
Krok 3 – Znaczenie biologiczne i poza wartością P (lub: czy to naprawdę miało wpływ na zwierzęta?)
W pewnym sensie łatwo jest zaburzyć strukturę społeczności mikrobiologicznej. Mikrobiota jelitowa, na przykład, jest podatna na zmiany w diecie, przestrzeni fizycznej i antybiotykach. Interwencje mogą łatwo spowodować istotne zmiany w składzie społeczności (zarówno różnorodności alfa jak i beta). W ostatnich dekadach przeprowadzono bardzo ważne prace, które to potwierdzają. Kluczowe są jednak dowody na znaczenie biologiczne. Wiele z "pracy" wykonywanej przez mikroby jest zbędna. Na przykład wielu członków mikrobioty jelitowej może produkować te same krótkołańcuchowe kwasy tłuszczowe. Dlatego zmiany w składzie społeczności mikrobów (czyli "kto tam jest") są prawdopodobnie mniej ważne niż to, co robią. Ważne jest, aby szukać pośrednich dowodów, że na to ostatnie wpłynęła interwencja, a to może przyjść na różne sposoby: tempo wzrostu, odporność na choroby, funkcja bariery jelitowej. To naprawdę zależy od badania, ale powinno być zgłoszone.
Jak w przypadku każdej innej najnowocześniejszej technologii, niektóre ważne aspekty mogą zostać utracone podczas przekładania badań z laboratorium na farmę. W miarę jak modulacja mikrobiomu i jej zastosowania w produkcji trzody chlewnej stają się coraz bardziej zrozumiałe, powinniśmy zobaczyć nowe strategie, które pomogą zwiększyć wydajność i odporność na choroby.





